Denne omfattende gjennomgangsrapporten er offisielt sendt til Eurosurveillance-redaksjonen den 27. november 2020 via deres innsendingsportal, vedlagt denne gjennomgangsrapporten er et brev om tilbaketrekking, signert av alle hoved- og medforfattere. Første og siste navn er de første og andre hovedforfatterne. Alle navn i mellom er medforfattere.
Pieter Borger(1), Bobby Rajesh Malhotra(2) , Michael Yeadon(3) , Clare Craig(4), Kevin McKernan(5) , Klaus Steger(6) , Paul McSheehy(7) , Lidiya Angelova(8), Fabio Franchi(9), Thomas Binder(10), Henrik Ullrich(11) , Makoto Ohashi(12), Stefano Scoglio(13), Marjolein Doesburg-van Kleffens(14), Dorothea Gilbert(15), Rainer Klement(16), Ruth Schruefer(17), Berber W. Pieksma(18), Jan Bonte(19), Bruno H. Dalle Carbonare(20), Kevin P. Corbett(21), Ulrike Kämmerer(22)
ABSTRAKT
I publikasjonen «Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR» (Eurosurveillance 25 (8) 2020) presenterer forfatterne en diagnostisk arbeidsflyt og RT-qPCR-protokoll for deteksjon og diagnostikk av 2019-nCoV (nå kjent som SARS-CoV-2), som de hevder å være validert, i tillegg til å være en robust diagnostisk metodikk for bruk i laboratorieinnstillinger for folkehelse.
I lys av alle konsekvensene som følger av denne publikasjonen for samfunn over hele verden, utførte en gruppe uavhengige forskere en punktvis gjennomgang av den nevnte publikasjonen der 1) alle komponentene i den presenterte testdesignen ble kryssjekket, 2) RT-qPCR-protokollanbefalinger ble vurdert god laboratoriepraksis, og 3) parametere undersøkt mot relevant vitenskapelig litteratur som dekker feltet.
Den publiserte RT-qPCR-protokollen for deteksjon og diagnostikk av 2019-nCoV og manuskriptet lider av mange tekniske og vitenskapelige feil, inkludert utilstrekkelig primer-design, en problematisk og utilstrekkelig RT-qPCR-protokoll og fravær av en nøyaktig testvalidering. Verken den presenterte testen eller manuskriptet i seg selv oppfyller kravene til en akseptabel vitenskapelig publikasjon. Videre nevnes ikke alvorlige interessekonflikter for forfatterne. Til slutt betyr den veldig korte tidsperioden mellom innsending og aksept av publikasjonen (24 timer) at en systematisk fagfellevurderingsprosess enten ikke ble utført her, eller at problematisk dårlig kvalitet var. Vi gir overbevisende bevis på flere vitenskapelige mangler, feil og mangler.
Med tanke på de vitenskapelige og metodiske skavanker som presenteres her, er vi sikre på at redaksjonen for Eurosurveillance ikke har annet valg enn å trekke publikasjonen tilbake.
KONSISSEVERSIKT RAPPORT
Denne artikkelen vil vise mange alvorlige feil i Corman-Drosten-papiret, hvis betydning har ført til verdensomspennende feildiagnostisering av infeksjoner tilskrevet SARS-CoV-2 og assosiert med sykdommen COVID-19. Vi står overfor strenge lockdowns som har ødelagt mange menneskers liv og levebrød, begrenset tilgang til utdanning og disse pålagte restriksjonene fra regjeringer rundt om i verden er et direkte angrep på folks grunnleggende rettigheter og deres personlige friheter, noe som resulterer i sikkerhetskader for hele økonomier på en global skala.
Det er ti fatale problemer med Corman-Drosten-papiret, som vi vil skissere og forklare mer detaljert i de følgende avsnittene.
Det første og store problemet er at romanen Coronavirus SARS-CoV-2 (i publikasjonen 2019-nCoV og i februar 2020 kalt SARS-CoV-2 av et internasjonalt konsortium av viruseksperter) er basert på silikonsekvenser (teoretiske) , levert av et laboratorium i Kina [1], fordi den gang var verken kontrollmateriale for smittsom («levende») eller inaktivert SARS-CoV-2 eller isolert genomisk RNA for viruset tilgjengelig for forfatterne. Hittil har ingen validering blitt utført av forfatterskapet basert på isolerte SARS-CoV-2-virus eller full lengde RNA derav. I følge Corman et al .:
«Vi hadde som mål å utvikle og distribuere robust diagnostisk metode for bruk i folkehelselaboratorium uten å ha virusmateriale tilgjengelig.» [1]
Fokus her bør plasseres på de to angitte målene: a) utvikling og b) distribusjon av en diagnostisk test for bruk i folkehelselaboratorium. Disse målene kan ikke oppnås uten å ha noe faktisk virusmateriale tilgjengelig (f.eks. For å bestemme den smittsomme virusmengden). Uansett kan bare en protokoll med maksimal nøyaktighet være det obligatoriske og primære målet i alle scenario-utfall av denne størrelsesorden. Kritisk bestemmelse av viral belastning er obligatorisk informasjon, og det er i Christian Drostens gruppeansvar å utføre disse eksperimentene og gi viktige data.
Likevel ble disse i silikosekvenser brukt til å utvikle en RT-PCR-testmetodikk for å identifisere det nevnte viruset. Denne modellen var basert på antagelsen om at det nye viruset er veldig likt SARS-CoV fra 2003, ettersom begge er beta-koronavirus.
PCR-testen ble derfor designet ved bruk av den genomiske sekvensen til SARS-CoV som kontrollmateriale for Sarbeco-komponenten; vi vet dette fra vår personlige e-postkommunikasjon med [2] en av medforfatterne av Corman-Drosten-papiret. Denne metoden for å modellere SARS-CoV-2 ble beskrevet i Corman-Drosten-papiret som følger:
«Etablering og validering av en diagnostisk arbeidsflyt for 2019-nCoV-screening og spesifikk bekreftelse, designet i fravær av tilgjengelige virusisolater eller originale pasientprøver. Design og validering ble muliggjort av den nære genetiske slekten til SARS-CoV fra 2003, og hjulpet av bruk av syntetisk nukleinsyre-teknologi. «
Omvendt transkripsjon-polymerase kjedereaksjon (RT-PCR) er en viktig biomolekylær teknologi for raskt å oppdage sjeldne RNA-fragmenter, som er kjent på forhånd. I det første trinnet transkriberes RNA -molekyler som er tilstede i prøven for å gi cDNA. CDNA blir deretter amplifisert i polymerasekjedereaksjonen ved bruk av et spesifikt primerpar og et termostabilt DNA -polymeraseenzym. Teknologien er svært sensitiv og dens påvisningsgrense er teoretisk sett 1 molekyl cDNA. Spesifisiteten til PCR er sterkt påvirket av biomolekylære designfeil.
Hva er viktig når du designer en RT-PCR-test og den kvantitative RT-qPCR-testen beskrevet i Corman-Drosten-publikasjonen?
Primerne og sonderne: a) konsentrasjonen av primere og prober må ha et optimalt område
(100-200 nM)
b) må være spesifikk for målgenet du vil forsterke
c) må ha en optimal prosentandel av GC -innhold i forhold til de totale nitrogenholdige basene (minimum 40%, maksimum 60%)
d) for virusdiagnostikk må minst 3 primerpar påvise 3 virale gener (helst så langt fra hverandre som mulig i virusgenomet)- Temperaturen der alle reaksjonene finner sted: a) DNA -smeltetemperatur (> 92 °)
b) DNA -amplifikasjonstemperatur (TaqPol -spesifikk)
c) Tm; glødetemperaturen (temperaturen der primerne og prober når målbindingen/løsrivelsen, ikke overstige 2 ̊C per primerpar). Tm avhenger sterkt av GC -innholdet i primerne - Antall forsterkningssykluser (mindre enn 35; fortrinnsvis 25-30 sykluser); Ved virusdeteksjon oppdager> 35 sykluser bare signaler som ikke korrelerer med smittsomt virus, bestemt ved isolering i cellekultur [gjennomgått i 2]; hvis noen blir testet av PCR som positiv når en terskel på 35 sykluser eller høyere brukes (som er tilfellet i de fleste laboratorier i Europa og USA), er sannsynligheten for at personen faktisk er infisert mindre enn 3%, sannsynligheten for at sa resultatet er et falskt positivt er 97% [anmeldt i 3]
- Molekylærbiologiske valideringer; amplifiserte PCR -produkter må valideres enten ved å kjøre produktene i en gel med en DNA -linjal, eller ved direkte DNA -sekvensering
- Positive og negative kontroller bør spesifiseres for å bekrefte/avkrefte spesifikk virusoppdagelse
- Det bør være en standard operasjonell prosedyre (SOP) tilgjengelig SOP spesifiserer parametrene ovenfor utvetydig, slik at alle laboratorier kan sette opp nøyaktig de samme testbetingelsene. For å ha en validert universell SOP er avgjørende, fordi den muliggjør sammenligning av data i og mellom land.
Mindre bekymringer med papiret CORMAN-DROSTEN
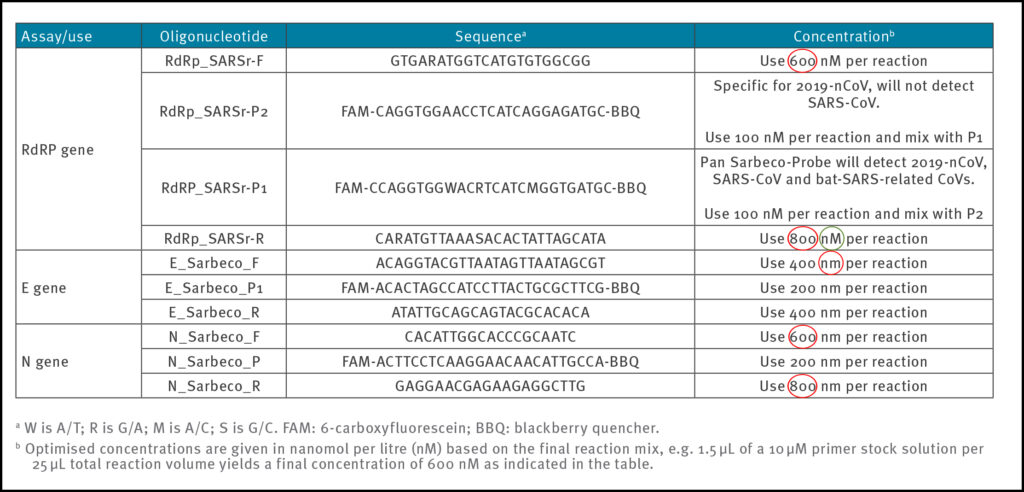
- I tabell 1 i Corman-Drosten-papiret er det angitt forskjellige forkortelser-«nM» er spesifisert, «nm» er ikke. Når det gjelder korrekt nomenklatur, betyr nm «nanometer», derfor bør nm lese nM her.
- Det er den generelle konsensus å skrive genetiske sekvenser alltid i 5’-3 ’retning, inkludert omvendte primere. Det er svært uvanlig å justere med omvendt komplementær skriving av primersekvensen slik forfatterne gjorde i figur 2 i Corman-Drosten-papiret. I tillegg er en wobble -base merket som «y» uten beskrivelse av basene Y står for.
- To villedende fallgruver i Corman-Drosten-papiret er at tabell 1 ikke inneholder Tm-verdier (glødetemperaturverdier), og det viser heller ikke GC-verdier (antall G og C i sekvensene som %-verdi av totale baser).
STORE bekymringer med KORMAN-DROSTEN-PAPIRET
A) BAKGRUNN
Forfatterne introduserer bakgrunnen for sitt vitenskapelige arbeid som: «Det pågående utbruddet av det nylig oppståtte nye koronaviruset (2019-nCoV) utgjør en utfordring for folkehelselaboratorier ettersom virusisolater ikke er tilgjengelige mens det er økende bevis på at utbruddet er mer utbredt enn opprinnelig tenkt, og internasjonal spredning gjennom reisende skjer allerede ”.
I følge BBC News [4] og Google Statistics [5] var det 6 dødsfall verden over 21. januar 2020-dagen da manuskriptet ble sendt. Hvorfor antok forfatterne en utfordring for folkehelselaboratorier mens det ikke var vesentlig bevis på den tiden som indikerte at utbruddet var mer utbredt enn først antatt?
Som et mål erklærte forfatterne å utvikle og distribuere robust diagnostisk metode for bruk i folkehelselaboratorium uten å ha virusmateriell tilgjengelig. Videre erkjenner de at «Denne studien demonstrerer den enorme responskapasiteten som er oppnådd gjennom koordinering av akademiske og offentlige laboratorier i nasjonale og europeiske forskningsnettverk.»
B) METODER OG RESULTATER
- Primer & Probe Design 1a) Feil primerkonsentrasjoner Pålitelige og nøyaktige PCR-testprotokoller er vanligvis designet med mellom 100 nM og 200 nM per primer [7]. I Corman-Drosten-papiret observerer vi uvanlig høye og varierende primerkonsentrasjoner for flere primere (tabell 1). For RdRp_SARSr-F og RdRp_SARSr-R primerpar er henholdsvis 600 nM og 800 nM beskrevet. Tilsvarende, for N_Sarbeco_F og N_Sarbeco_R primersettet, anbefaler de henholdsvis 600 nM og 800 nM [1]. Det bør være klart at disse konsentrasjonene er altfor høye til å være optimale for spesifikke forsterkninger av målgener. Det finnes ingen spesifisert grunn til å bruke disse ekstremt høye konsentrasjonene av primere i denne protokollen. Disse konsentrasjonene fører heller til økt uspesifikk binding og PCR -produktforsterkning.
1b) Uspesifiserte (“wobbly”) primer- og probesekvenser
For å oppnå reproduserbare og sammenlignbare resultater, er det avgjørende å definere primerparene tydelig. I Corman-Drosten-papiret observerte vi seks uspesifiserte posisjoner, angitt med bokstavene R, W, M og S (tabell 2). Bokstaven W betyr at i denne posisjonen kan det være enten A eller T; R betyr at det kan være enten G eller A; M indikerer at posisjonen enten kan være A eller C; bokstaven S indikerer at det kan være enten G eller C på denne posisjonen.
Dette store antallet varianter er ikke bare uvanlig, men det er også svært forvirrende for laboratorier. Disse seks uspesifiserte posisjonene kan lett resultere i design av flere forskjellige alternative primersekvenser som ikke er relatert til SARS-CoV-2 (2 distinkte RdRp_SARSr_F primere + 8 distinkte RdRp_SARS_P1 sonder + 4 distinkte RdRp_SARSr_R). Designvariasjonene vil uunngåelig føre til resultater som ikke engang er SARS CoV-2-relaterte. Derfor er den forvirrende uspesifikke beskrivelsen i Corman-Drosten-papiret ikke egnet som en standard operasjonell protokoll. Disse uspesifiserte posisjonene burde vært utformet utvetydig.
Disse vaklende sekvensene har allerede skapt en bekymring i feltet og resulterte i et brev til redaktøren forfattet av Pillonel et al. [8] angående åpenbare feil i de beskrevne sekvensene. Disse feilene er åpenbare i Corman et al. supplement også.
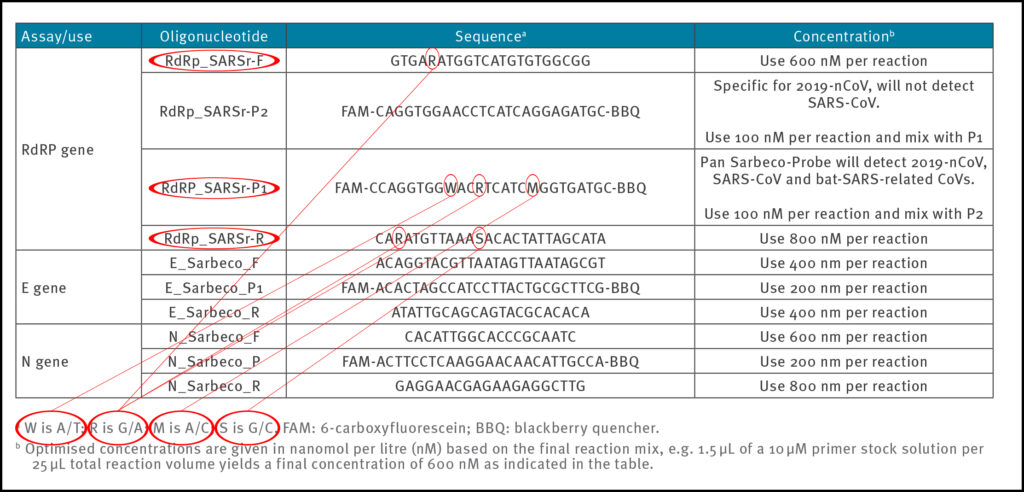
WHO-protokollen (figur 1), som stammer direkte fra Corman-Drosten-papiret, konkluderer med at to kontrollgener (E- og RdRp-genene) må identifiseres for å bekrefte tilstedeværelsen av SARS-CoV-2 i analysen. Det skal bemerkes at RdRd-genet har en usikker posisjon («vinglet») i fremover-primeren (R = G/A), to usikre posisjoner i revers-primeren (R = G/A; S = G /C) og den har tre usikre posisjoner i RdRp-sonden (W = A/T; R = G/A; M = A/C). Så to forskjellige fremoverprimere, fire forskjellige reversprimere og åtte forskjellige sonder kan syntetiseres for RdRd-genet. Til sammen er det 64 mulige kombinasjoner av primere og sonder!
Corman-Drosten-papiret identifiserer videre et tredje gen som ifølge WHO-protokollen ikke ble ytterligere validert og ansett som unødvendig:
«Vær oppmerksom på at N -genanalysen også fungerte bra, men ble ikke utsatt for intensiv ytterligere validering fordi den var litt mindre sensitiv.»
Dette var et uheldig utelatelse, da det ville være best å bruke alle tre gen -PCR -ene som bekreftende analyser, og dette ville ha resultert i en tilstrekkelig tilstrekkelig virus -RNA -deteksjonsdiagnostisk verktøyprotokoll. Tre bekreftende analysetrinn vil i det minste minimere feil og usikkerheter ved hvert foldetrinn når det gjelder “Wobbly” -flekker. (Ikke desto mindre vil protokollen fortsatt mangle enhver «god laboratoriepraksis» når alle andre designfeil tas i betraktning).
Slik det er, er N-genanalysen dessverre verken foreslått i WHO-anbefalingen (figur 1) som et obligatorisk og avgjørende tredje bekreftende trinn, og det blir heller ikke understreket i Corman-Drosten-papiret som en viktig valgfri trygghet «for en rutinemessig arbeidsflyt» (Tabell 2).
Følgelig ble det i nesten alle testprosedyrer over hele verden bare brukt 2 primermatcher i stedet for alle tre. Dette tilsynet gjør hele testprotokollen ubrukelig når det gjelder å levere nøyaktige testresultater av reell betydning i en pågående pandemi.
Figur 1: N-Gene-bekreftelsesanalysen fremheves verken som nødvendig tredje trinn i den offisielle anbefalingen for Drosten-Corman-protokollen fra WHO nedenfor [8], og er heller ikke nødvendig som et avgjørende trinn for høyere testnøyaktighet i Eurosurveillance-publikasjonen.

1c) Feilaktig GC-innhold (omtalt i 2c, sammen med glødetemperatur (Tm))
1d) Påvisning av virale gener
RT-PCR anbefales ikke for primær diagnostikk av infeksjon. Dette er grunnen til at RT-PCR-testen som brukes i klinisk rutine for påvisning av COVID-19, ikke er indisert for diagnostisering av COVID-19 på forskriftsmessig grunnlag.
“Klinikere må anerkjenne den forbedrede nøyaktigheten og hastigheten til de molekylære diagnostiske teknikkene for diagnostisering av infeksjoner, men også for å forstå begrensningene deres. Laboratorieresultater bør alltid tolkes i sammenheng med den kliniske presentasjonen av pasienten, og passende sted, kvalitet og tidspunkt for prøvetaking er nødvendig for pålitelige testresultater ”. [9]
Den kan imidlertid brukes til å hjelpe legens differensialdiagnose når han eller hun må skille mellom forskjellige infeksjoner i lungen (influensa, Covid-19 og SARS har svært like symptomer). For en bekreftende diagnose av et spesifikt virus må minst 3 spesifikke primerpar påføres for å påvise 3 virusspesifikke gener. Fortrinnsvis bør disse målgenene være lokalisert med størst mulig avstand i virusgenomet (motsatte ender inkludert).
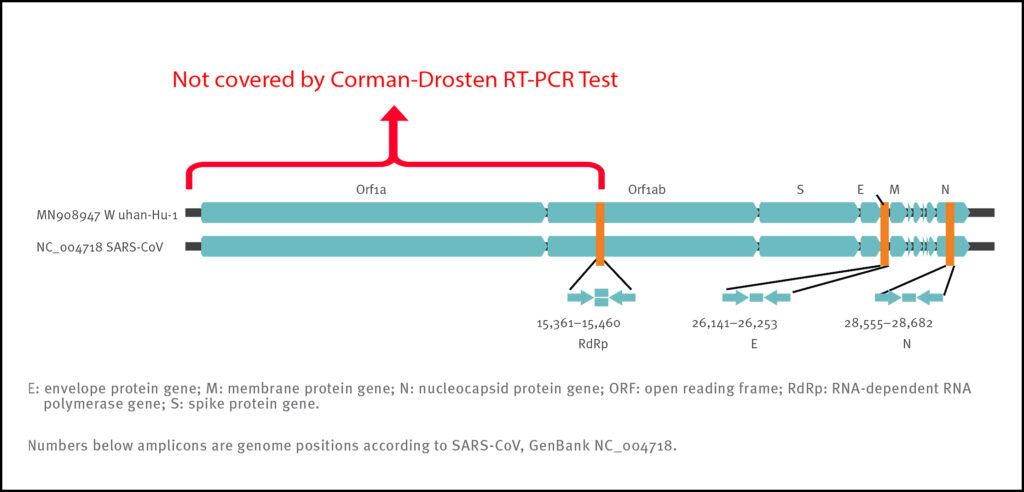
Selv om Corman-Drosten-papiret beskriver 3 primere, dekker disse primerne bare omtrent halvparten av virusets genom. Dette er en annen faktor som reduserer spesifisiteten for påvisning av intakt COVID-19 virus-RNA og øker sitatet for falske positive testresultater.
Derfor, selv om vi får tre positive signaler (dvs. de tre primerparene gir 3 forskjellige forsterkningsprodukter) i en prøve, beviser ikke dette tilstedeværelsen av et virus. En bedre primer -design ville ha terminale primere i begge ender av virusgenomet. Dette er fordi hele virusgenomet ville bli dekket og tre positive signaler bedre kan skille mellom et komplett (og dermed potensielt smittsomt) virus og fragmenterte virale genomer (uten smittsom potens). For å utlede noe av betydning om virusets smittsomhet, skulle Orf1-genet, som koder for det essensielle replikaseenzymet til SARS-CoV-virus, ha blitt inkludert som et mål (figur 2). Plasseringen av målene i regionen av virusgenomet som er mest tungt og variabelt transkribert er en annen svakhet ved protokollen.
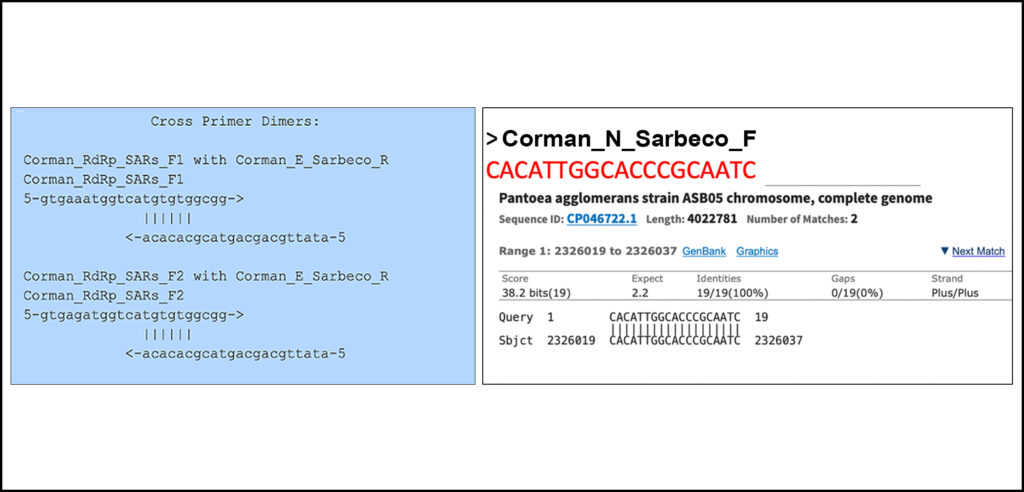
Kim et al. demonstrere et svært variabelt 3′-uttrykk for subgenomisk RNA i Sars-CoV-2 [23]. Disse RNA-ene overvåkes aktivt som signaturer for asymptomatiske og ikke-smittsomme pasienter [10]. Det er svært tvilsomt å skjerme en populasjon av asymptomatiske mennesker med qPCR-primere som har 6 basepar primer-dimer på den 3 primære enden av en primer (figur 3).
Tilsynelatende anbefaler WHO disse primerne. Vi testet alle wobble-derivatene fra Corman-Drosten-papiret med Thermofishers primer dimer webverktøy [11]. RdRp forover -primeren har 6bp 3prime -homologi med Sarbeco E Reverse. Ved høye primerkonsentrasjoner er dette nok til å skape unøyaktigheter.
Merk: Det er en perfekt match av en av N-primerne til et klinisk patogen (Pantoea), funnet hos immunkompromitterte pasienter. Den omvendte primeren treffer også Pantoea, men ikke i samme region (figur 3).
Dette er alvorlige designfeil, siden testen ikke kan skille mellom hele viruset og virusfragmentene. Testen kan ikke brukes som diagnostikk for SARS-virus.
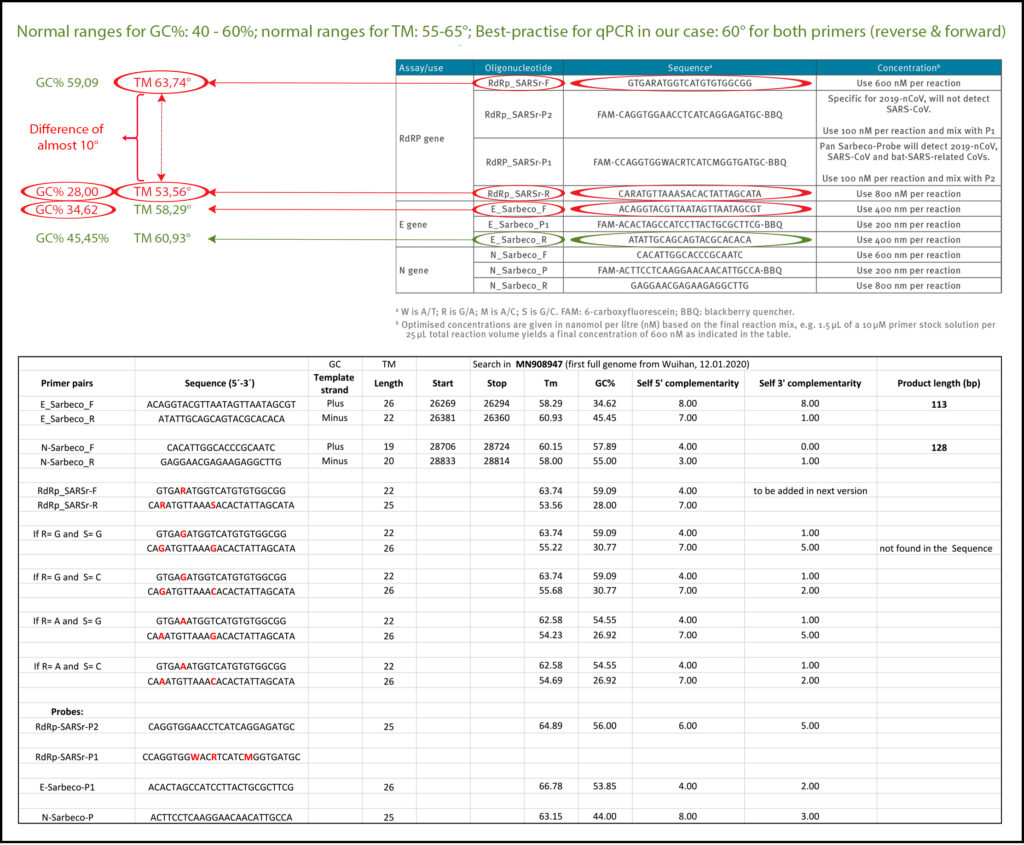
Reaksjonstemperaturer 2a) DNA -smeltetemperatur (> 92 °). Tilstrekkelig adressert i Corman-Drosten-papiret. 2b) DNA -amplifikasjonstemperatur. Tilstrekkelig adressert i Corman-Drosten-papiret. 2c) Feilaktig GC-innhold og Tm Glødningstemperaturen bestemmer ved hvilken temperatur primeren fester/løsner fra målsekvensen. For en effektiv og spesifikk forsterkning bør GC -innholdet i primere oppfylle minst 40% og maksimalt 60% forsterkning. Som angitt i tabell 3, er tre av primerne beskrevet i Corman-Drosten-papiret ikke innenfor det normale området for GC-innhold. To primere (RdRp_SARSr_F og RdRp_SARSr_R) har uvanlige og svært lave GC-verdier på 28% -31% for alle mulige varianter av wobble baser, mens primer E_Sarbeco_F har en GC-verdi på 34,6% (tabell 3 og andre panel i tabell 3) . Det skal bemerkes at GC-innholdet i stor grad bestemmer bindingen til dets spesifikke mål på grunn av dets tre hydrogenbindinger i baseparring. Jo lavere GC-innhold i primeren er, desto lavere er bindingsevnen til dens spesifikke målgen-sekvens (dvs. genet som skal detekteres). Dette betyr at for at en målsekvens skal gjenkjennes må vi velge en temperatur som er så nær som mulig den faktiske glødingstemperaturen (beste praksis-verdi) for at primeren ikke skal løsne igjen, samtidig som vi spesifikt velger målsekvens. Hvis Tm-verdien er veldig lav, som observert for alle wobbly-varianter av RdRp revers primere, kan primerne binde ikke-spesifikt til flere mål, redusere spesifisiteten og øke potensielle falske positive resultater. Glødningstemperaturen (Tm) er en avgjørende faktor for bestemmelsen av spesifisiteten/nøyaktigheten til qPCR-prosedyren og avgjørende for å evaluere nøyaktigheten til qPCR-protokoller. Anbefaling om beste praksis: Både primere (forover og bakover) bør ha en nesten lik verdi, helst den samme verdien. Vi brukte den fritt tilgjengelige primer-designprogramvaren Primer-BLAST [12, 25] for å evaluere beste praksisverdier for alle primere som ble brukt i Corman-Drosten-papiret (tabell 3). Vi forsøkte å finne en Tm-verdi på 60 ° C, mens vi på samme måte søkte høyest mulig GC%-verdi for alle primere. En maksimal Tm -forskjell på 2 ° C i primerpar ble ansett som akseptabel. Ved å teste primerparene som er spesifisert i Corman-Drosten-papiret, observerte vi en forskjell på 10 ° C med hensyn til glødetemperaturen Tm for primerpar1 (RdRp_SARSr_F og RdRp_SARSr_R). Dette er en veldig alvorlig feil og gjør protokollen ubrukelig som et spesifikt diagnostisk verktøy. Ytterligere testing viste at bare primerparet designet for å amplifisere N-genet (N_Sarbeco_F og N_Sarbeco_R) nådde tilstrekkelig standard for å operere i en diagnostisk test, siden det har et tilstrekkelig GC-innhold og Tm-forskjellen mellom primerne (N_Sarbeco_F og N_Sarbeco_R ) er 1,85 ° C (under den avgjørende maksimumsgrensen på 2 ° C). Viktigere er dette genet som verken ble testet i virusprøvene (tabell 2) eller understreket som en bekreftende test. I tillegg til svært varierende smeltetemperaturer og degenererte sekvenser i disse primerne, er det en annen faktor som påvirker spesifisiteten til prosedyren: dNTP -ene (0,4 uM) er 2x høyere enn anbefalt for en høyspesifikk forsterkning. Det er også tilsatt magnesiumsulfat til reaksjonen. Denne prosedyren kombinert med en lav glødetemperatur kan skape uspesifikke forsterkninger. Når ytterligere magnesium er nødvendig for qPCR, bør analysens spesifisitet undersøkes nærmere. Designfeilene som er beskrevet her er så alvorlige at det er svært lite sannsynlig at spesifikk amplifikasjon av SARS-CoV-2 genetisk materiale vil skje ved bruk av protokollen til Corman-Drosten-papiret.
Antall forsterkningssykluser Det skal bemerkes at det ikke er nevnt noe sted i Corman-Drosten-papiret at en test er positiv eller negativ, eller faktisk hva som definerer et positivt eller negativt resultat. Disse typer virologiske diagnostiske tester må være basert på en SOP, inkludert et validert og fast antall PCR -sykluser (Ct -verdi), hvoretter en prøve anses som positiv eller negativ. Den maksimalt rimelige Ct -verdien er 30 sykluser. Over en Ct på 35 sykluser må det forventes raskt økende antall falske positiver. PCR -data evaluert som positive etter en Ct -verdi på 35 sykluser er helt upålitelige. Siterer Jaafar et al. 2020 [3]: «Ved Ct = 35, verdien vi brukte for å rapportere et positivt resultat for PCR, er <3% av kulturene positive.» Med andre ord var det ingen vellykket virusisolasjon av SARS-CoV-2 ved de høye Ct-verdiene. Videre viser vitenskapelige studier at bare ikke-smittsomme (døde) virus påvises med Ct-verdier på 35 [22]. Mellom 30 og 35 er det et grått område, hvor en positiv test ikke kan fastslås med sikkerhet. Dette området bør utelukkes. Selvfølgelig kan man utføre 45 PCR-sykluser, som anbefalt i Corman-Drosten WHO-protokollen (figur 4), men da må du også definere en rimelig Ct-verdi (som ikke bør overstige 30). Men et analytisk resultat med en Ct-verdi på 45 er vitenskapelig og diagnostisk absolutt meningsløst (en rimelig Ct-verdi bør ikke overstige 30). Alt dette bør kommuniseres veldig tydelig. Det er en betydelig feil at Corman-Drosten-papiret ikke nevner maksimal Ct-verdi som en prøve utvetydig kan betraktes som et positivt eller negativt testresultat. Denne viktige syklusterskelgrensen er heller ikke spesifisert i noen oppfølgingsinnleveringer til dags dato.

- Biomolekylære valideringer For å avgjøre om de forsterkede produktene faktisk er SARS-CoV-2-gener, er biomolekylær validering av amplifiserte PCR-produkter avgjørende. For en diagnostisk test er denne validering et absolutt must. Validering av PCR-produkter bør utføres ved enten å kjøre PCR-produktet i en 1% agarose-EtBr-gel sammen med en størrelsesindikator (DNA-linjal eller DNA-stige) slik at størrelsen på produktet kan estimeres. Størrelsen må tilsvare den beregnede størrelsen på forsterkningsproduktet. Men det er enda bedre å sekvensere forsterkningsproduktet. Sistnevnte vil gi 100% sikkerhet om identiteten til forsterkningsproduktet. Uten molekylær validering kan man ikke være sikker på identiteten til de forsterkede PCR -produktene. Med tanke på de alvorlige designfeilene som er beskrevet tidligere, kan de forsterkede PCR -produktene være hva som helst. Heller ikke nevnt i Corman-Drosten-papiret er tilfellet med små fragmenter av qPCR (rundt 100 bp): Det kan enten være 1,5% agarosegel eller til og med en akrylamidgel. Det faktum at disse PCR-produktene ikke har blitt validert på molekylært nivå er en annen slående feil i protokollen, noe som gjør enhver test basert på den ubrukelig som et spesifikt diagnostisk verktøy for å identifisere SARS-CoV-2-viruset.
- Positive og negative kontroller for å bekrefte/tilbakevise spesifikk virusoppdagelse. Den ubekreftede antagelsen beskrevet i Corman-Drosten-papiret er at SARS-CoV-2 er det eneste viruset fra den SARS-lignende beta-coronavirus-gruppen som for tiden forårsaker infeksjoner hos mennesker. Sekvensene som deres PCR-metode er basert på, er i silikosekvenser, levert av et laboratorium i Kina [23], fordi det på tidspunktet for utviklingen av PCR-testen ikke var noe kontrollmateriale for smittsomme (“levende”) eller inaktiverte SARS-CoV- 2 var tilgjengelig for forfatterne. PCR-testen ble derfor designet ved å bruke sekvensen til det kjente SARS-CoV som kontrollmateriale for Sarbeco-komponenten (Dr. Meijer, medforfatter av Corman-Drosten-papir i en e-postutveksling med Dr. Peter Borger) [2]. Alle individer som tester positivt med RT-PCR-testen, som beskrevet i Corman-Drosten-papiret, antas å være positive for SARS-CoV-2-infeksjoner. Det er tre alvorlige feil i deres antagelse. For det første kan en positiv test for RNA-molekylene beskrevet i Corman-Drosten-papiret ikke sidestilles med «infeksjon med et virus». En positiv RT-PCR-test indikerer bare tilstedeværelsen av virale RNA-molekyler. Som vist under punkt 1d (ovenfor), var ikke Corman-Drosten-testen designet for å påvise viruset i full lengde, men bare et fragment av viruset. Vi har allerede konkludert med at dette klassifiserer testen som uegnet som en diagnostisk test
for SARS-virusinfeksjoner. For det andre og av stor relevans, ble ikke funksjonaliteten til den publiserte RT-PCR-testen demonstrert ved bruk av en positiv kontroll (isolert SARS-CoV-2 RNA) som er en vesentlig vitenskapelig gullstandard. For det tredje sier Corman-Drosten-papiret: «For å vise at analysene kan påvise andre flaggermus-assosierte SARS-relaterte virus, brukte vi E-genanalysen til å teste seks flaggermus-avledede avføringsprøver tilgjengelig fra Drexler et al. […] Og Muth et al. […]. Disse virus-positive prøvene stammer fra europeiske rhinolophid-flaggermus. Påvisning av disse fylogenetiske utfallene i den SARS-relaterte CoV-kladen antyder at alle asiatiske virus sannsynligvis vil bli oppdaget. Dette ville, teoretisk sett, sikre bred sensitivitet, selv ved flere uavhengige anskaffelser av variantvirus fra et dyrereservoar. ” Denne uttalelsen viser at E-genet som brukes i RT-PCR-test, som beskrevet i Corman-Drosten-papiret, ikke er spesifikt for SARS-CoV-2. E -genprimerne oppdager også et bredt spekter av andre SARS -virus.
Genomet til koronaviruset er det største av alle RNA -virus som infiserer mennesker, og de har alle en veldig lik molekylstruktur. Likevel har SARS-CoV1 og SARS-CoV-2 to svært spesifikke genetiske fingeravtrykk, som skiller dem fra de andre koronavirusene. For det første er en unik fingeravtrykkssekvens (KTFPPTEPKKDKKKK) tilstede i N-proteinet til SARS-CoV og SARS-CoV-2 [13,14,15]. For det andre inneholder både SARS-CoV1 og SARS-CoV2 ikke HE-proteinet, mens alle andre koronavirus har dette genet [13, 14]. Så, for å spesifikt oppdage et SARS-CoV1 og SARS-CoV-2 PCR-produkt, burde regionen ovenfor i N-genet blitt valgt som amplifikasjonsmål. En pålitelig diagnostisk test bør fokusere på denne spesifikke regionen i N -genet som en bekreftende test. PCR for dette N-genet ble ikke ytterligere validert eller anbefalt som et testgen av Drosten-Corman-papiret, på grunn av at den var «ikke så sensitiv» med den originale SARS-CoV-sonden [1].
Videre gjør fraværet av HE-genet i både SARS-CoV1 og SARS-CoV-2 dette genet den ideelle negative kontrollen for å utelukke andre koronavirus. Corman-Drosten-papiret inneholder ikke denne negative kontrollen, og det inneholder heller ingen andre negative kontroller. PCR-testen i Corman-Drosten-papiret inneholder derfor verken en unik positiv kontroll eller en negativ kontroll for å utelukke tilstedeværelsen av andre koronavirus. Dette er en annen stor designfeil som klassifiserer testen som uegnet for diagnose.
Standard driftsprosedyre (SOP) er ikke tilgjengelig Det bør være en standard driftsprosedyre (SOP) tilgjengelig, som entydig angir parametrene ovenfor, slik at alle laboratorier kan sette opp de samme testbetingelsene. For å ha en validert universell SOP er avgjørende, fordi det muliggjør datasammenligning i og mellom land. Det er veldig viktig å spesifisere alle primerparametere utvetydig. Vi merker oss at dette ikke er gjort. Ct -verdien for å indikere når en prøve skal betraktes som positiv eller negativ er ikke spesifisert. Det er heller ikke spesifisert når en prøve anses infisert med SARS-CoV-virus. Som vist ovenfor kan ikke testen skille mellom virus- og virusfragmenter, så Ct -verdien som indikerer positivitet er avgjørende viktig. Denne Ct-verdien burde vært spesifisert i Standard Operational Procedure (SOP) og lagt på nett slik at alle laboratorier som utfører denne testen har nøyaktig de samme grensebetingelsene. Det peker på feil vitenskap om at en slik SOP ikke eksisterer. Laboratoriene står dermed fritt til å gjennomføre testen som de finner passende, noe som resulterer i en enorm variasjon. Laboratorier over hele Europa sitter igjen med en rekke spørsmål; hvilke primere å bestille? hvilke nukleotider som skal fylles ut på de udefinerte stedene? hvilken Tm -verdi skal du velge? Hvor mange PCR -sykluser skal kjøres? Ved hvilken Ct -verdi er prøven positiv? Og når er det negativt? Og hvor mange gener som skal testes? Bør alle gener testes, eller bare E- og RpRd-genet som vist i tabell 2 i Corman-Drosten-papiret? Bør N -genet testes også? Og hva er deres negative kontroll? Hva er deres positive kontroll? Protokollen som beskrevet er dessverre veldig vag og feilaktig i utformingen at man kan gå i dusinvis av forskjellige retninger. Det ser ikke ut til å være noen standardisering eller SOP, så det er ikke klart hvordan denne testen kan implementeres.
Konsekvenser av feilene beskrevet under 1-5: falske positive resultater. RT-PCR-testen beskrevet i Corman-Drosten-papiret inneholder så mange molekylærbiologiske designfeil (se 1-5) at det ikke er mulig å oppnå entydige resultater. Det er uunngåelig at denne testen vil generere et enormt antall såkalte «falske positiver». Definisjonen av falske positiver er en negativ prøve, som i utgangspunktet scorer positivt, men som er negativ etter ny test med samme test. Falske positive er feil positive testresultater, dvs. negative prøver som tester positive. Og dette er faktisk det som finnes i Corman-Drosten-papiret. På side 6 i manuskriptet PDF viser forfatterne at selv under godt kontrollerte laboratorieforhold genereres en betydelig prosentandel av falske positiver med denne testen: «I fire individuelle testreaksjoner ble svak initial reaktivitet sett, men de var negative ved ny test med samme analyse. Disse signalene var ikke assosiert med et bestemt virus, og for hvert virus som oppsto første positive reaktivitet, var det andre prøver som inneholdt det samme viruset ved en høyere konsentrasjon, men som ikke testet positivt. Gitt resultatene fra den omfattende tekniske kvalifikasjonen beskrevet ovenfor, ble det konkludert med at denne første reaktiviteten ikke skyldtes kjemisk ustabilitet i sanntid PCR-sonder og mest sannsynlig håndtering av problemer forårsaket av rask introduksjon av nye diagnostiske tester og kontroller under denne evalueringen studere.» [1] Den første setningen i dette utdraget er klare bevis på at PCR-testen beskrevet i Corman-Drosten-papiret genererer falske positiver. Selv under de godt kontrollerte forholdene i det toppmoderne Charité-laboratoriet er 4 av 310 primærtester falske positiver per definisjon. Fire negative prøver ble først testet positive, deretter negative når de ble testet på nytt. Dette er det klassiske eksempelet på en falsk positiv. I dette tilfellet identifiserer forfatterne dem ikke som falske positiver, noe som er intellektuelt uærlig. En annen avslørende observasjon i utdraget ovenfor er at forfatterne forklarer de falske positivene bort som «håndtering av problemer forårsaket av rask introduksjon av nye diagnostiske tester». Tenk deg laboratoriene som må innføre testen uten all nødvendig informasjon som normalt er beskrevet i en SOP.
Corman-Drosten-papiret ble ikke fagfellevurdert Før formell publisering i et vitenskapelig tidsskrift, er vitenskapelige og medisinske artikler tradisjonelt sertifisert av «fagfellevurdering». I denne prosessen tar tidsskriftets redaktører råd fra forskjellige eksperter («dommere») som har vurdert oppgaven og kan identifisere svakheter i antagelsene, metodene og konklusjonene. Vanligvis vil et tidsskrift bare publisere en artikkel når redaktørene er fornøyd med at forfatterne har tatt opp dommernes bekymringer og at dataene som presenteres støtter konklusjonene i avisen. ” Denne prosessen er like godt beskrevet for Eurosurveillance [16]. Corman-Drosten-papiret ble sendt til Eurosurveillance 21. januar 2020 og godkjent for publisering 22. januar 2020. 23. januar 2020 var avisen online. 13. januar 2020 ble versjon 1-0 av protokollen publisert på det offisielle WHO-nettstedet [17], oppdatert 17. januar 2020 som dokumentversjon 2-1 [18], selv før Corman-Drosten-papiret ble publisert 23. januar kl. Euroovervåkning. Normalt er fagfellevurdering en tidkrevende prosess siden minst to eksperter fra feltet må lese og kommentere det innsendte papiret kritisk. Etter vår mening var denne artikkelen ikke fagfellevurdert. Tjuefire timer er rett og slett ikke nok til å gjennomføre en grundig fagfellevurdering. Konklusjonen vår støttes av det faktum at et stort antall svært alvorlige designfeil ble funnet av oss, noe som gjør PCR-testen helt uegnet som et diagnostisk verktøy for å identifisere SARS-CoV-2-viruset. Enhver molekylærbiolog som er kjent med RT-PCR-design, ville lett ha observert de alvorlige feilene i Corman-Drosten-papiret før selve gjennomgangsprosessen. Vi ba Eurosurveillance 26. oktober 2020 om å sende oss en kopi av fagfellevurderingsrapporten. Til dags dato har vi ikke mottatt denne rapporten, og i et brev av 18. november 2020 nektet ECDC som vert for Eurosurveillance å gi tilgang uten å gi vesentlige vitenskapelige årsaker til beslutningen. Tvert imot skriver de at «avsløring ville undergrave formålet med vitenskapelige undersøkelser.» [24].
Leave a Reply
Du må være innlogget for å kunne kommentere.